Artigo apresentado originalmente no EATI – Encontro Anual de Tecnologia da Informação, sob o título “Sistema Inteligente para Apoio ao Diagnóstico de Diabetes Empregando Redes Neurais”.
Feito por cinco autores*.
***
As Redes Neurais Artificiais (RNAs) são um paradigma de processamento de informação inspirado no sistema neural biológico, o cérebro humano. As RNAs são sistemas massivamente paralelos e distribuídos, formados por unidades de processamento simples, que calculam determinadas funções, normalmente não lineares. Essas unidades são distribuídas em camadas, sendo interligadas por conexões, as quais se associam a pesos, que armazenam o conhecimento representado na rede, servindo para ponderar as entradas recebidas por unidade constituinte (Haykin, 2001).
A capacidade de aprender por meio de exemplos e de generalizar a informação aprendida é, sem dúvida, o principal atrativo da solução de problemas por meio de RNAs. As RNAs são aptas a resolver problemas de cunho geral, tais como aproximação, classificação, categorização e predição, entre outros (Braga et. al., 2000) (Lorenzi; Silveira, 2011). Nesse contexto, uma das aplicações das RNAs é apoiar o diagnóstico de doenças, classificando os pacientes como portadores ou não de uma determinada enfermidade.
O trabalho aqui apresentado aplicou, como estudo de caso, o diagnóstico de diabetes, doença cuja incidência tem aumentado rapidamente em nível mundial. Recentemente, a Organização Mundial de Saúde (OMS) reconheceu que a doença é epidêmica. As estatísticas apontam que o número de casos, em todo o mundo, atualmente chega a 246 milhões. Até 2025, esse número deve chegar a 350 milhões, de acordo com a Federação Internacional de Diabetes (IDF). No Brasil, segundo dados do Ministério da Saúde, estima-se que existam aproximadamente 11 milhões de portadores de diabetes, sendo que 7,5 milhões já sabem que têm a doença (Oliveira; Vencio, 2014). Embora a detecção de diabetes esteja melhorando, o tempo para diagnosticá-lo pode ser superior a 10 anos, a contar do início da doença até a concretização do diagnóstico.
Visando a auxiliar os profissionais da área de saúde, podem ser utilizados sistemas computacionais com a finalidade de apoiar o diagnóstico. Tais sistemas processam informações, com mais detalhe e em menor tempo, quando comparados com aos seres humanos, proporcionando uma melhora na qualidade dos serviços médicos, além de contribuir para a difusão de conhecimentos especializados (Kayaer; Yıldırım, 2003).
Nesse sentido, desenvolvemos um protótipo de sistema inteligente, empregando Redes Neurais multicamadas, para apoiar o diagnóstico de diabetes: o Sistema Neural para Apoio ao Diagnóstico de Diabetes (SND).
Para treinar e testar a aplicação da RNA no SND foi utilizada a base de dados da Universidade da Califórnia (UCI, 2014), denominada Pima Indians Diabetes (PID). As informações para compor essa base foram coletadas na comunidade indígena Pima, que vive perto de Phoenix, Arizona, Estados Unidos. Todas as pessoas cujos dados foram coletados são mulheres, com idade igual ou superior a 21 anos.
Diversas pesquisas foram realizadas nessa comunidade, pois ela é conhecida por apresentar a maior taxa de incidência de diabetes do mundo. Entre os adultos, 50% são diabéticos, além de apresentarem uma alta prevalência de obesidade (Baier e Hanson, 2004).
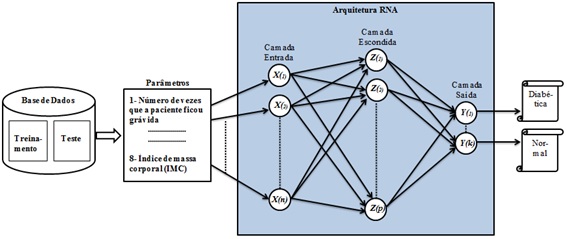
O Sistema Neural para Apoio ao Diagnóstico de Diabetes (SND) implementado tem como pilar computacional a RNA feedforward, com múltiplas camadas, cujo treinamento é supervisionado utilizando o algoritmo de treinamento back-propagation. Para desenvolver o sistema, foi utilizada a linguagem de programação Java, juntamente com o Sistema Gerenciador de Banco de Dados SQL Server. A figura 1 apresenta a arquitetura de alto nível do SND.
A validação do SND foi realizada com o subconjunto definido para a etapa de teste, sendo que essas informações, embora pertencentes ao PID, eram até então desconhecidas pelo sistema. O total de pacientes usados para compor o teste foi 230 – destes, 70% são classificados como normais e 30%, diabéticos.
A arquitetura da RNA utilizada possui uma camada de entrada, com oito neurônios, e uma de saída, com dois neurônios. O número de neurônios na camada de entrada é igual à quantidade de informações utilizadas para classificar cada paciente, ou seja, oito. Os neurônios da camada oculta (ou camada escondida) foram obtidos de forma empírica. Já os da camada saída foram fixados de acordo com as possíveis classificações para cada paciente (diabético ou normal), ou seja, dois.
O sistema proposto realizou 2.262 iterações para chegar aos resultados, obtendo uma taxa de acerto de 81,31%, o que resultou em 187 acertos e 43 erros (18,69%). Esse resultado torna o sistema muito promissor, principalmente se comparado aos resultados obtidos pelos trabalhos apresentados em Polat (Polat et. al., 2008) e Kayaer e Yildirim (2003).
Referências
- BAIER, L. J.; HANSON, R. L. Genetic studies of the etiology of type 2 diabetes in Pima Indians. Diabetes, 53, 1181–1186, 2004.
- BRAGA, A. P.; CARVALHO, A. C. P. L. F.; LUDERMIR, T. B. Redes Neurais Artificiais: teoria e aplicações. Rio de Janeiro: Livros Técnicos e Científicos, 2000.
- HAYKIN, Simon. Redes Neurais: princípios e prática. 2 ed. Porto Alegre: Bookman, 2001.
- KAYAER, K.; YILDIRIM T. Medical diagnosis on Pima Indian diabetes using general regression neural networks. In: Proceedings of the International Conference on Artificial Neural Networks and Neural Information Processing (ICANN/ICONIP) (pp. 181–184), 2003.
- LORENZI, F.; SILVEIRA, S. R. Desenvolvimento de Sistemas de Informação Inteligentes. Porto Alegre: UniRitter, 2011.
- OLIVEIRA, J. E. P.; VENCIO, S. Diretrizes da Sociedade Brasileira de Diabetes. São Paulo: AC Farmacêutica, 2014.
- POLAT, K.; GUNES, S.; ASLAN, A. A cascade learning system for classification of diabetes disease: Generalized discriminant analysis and least square support vector machine. Expert Systems with Applications, Volume 34, Issue 1, January 2008, Pages 482-487.
- UCI. University of California, Machine Learning and Intelligent System, School of Information and Computer Science, 2010. Acessado em: 17/08/2014 https://archive.ics.uci.edu/ml/datasets/Pima+Indians+Diabetes
*Autores:
- Maik Basso, Graduando em Sistemas de Informação (UFSM – Universidade Federal de Santa Maria/Campus de Frederico Westphalen-RS), maik@maikbasso.com.br.
- João Paulo Vieira, Graduando em Sistemas de Informação ( UFSM – Universidade Federal de Santa Maria/Campus de Frederico Westphalen-RS), vieira.jpviera@gmail.com.
- Fábio José Parreira, Doutor em Engenharia Elétrica (UFU – Universidade Federal de Uberlândia), Professor Associado do Departamento de Tecnologia da Informação (UFSM – Universidade Federal de Santa Maria/Campus de Frederico Westphalen-RS), fabiojparreira@gmail.com.
- Sidnei Renato Silveira, Doutor em Ciência da Computação (UFRGS – Universidade Federal do Rio Grande do Sul), Professor Adjunto do Departamento de Tecnologia da Informação (UFSM – Universidade Federal de Santa Maria/Campus de Frederico Westphalen-RS), sidneirenato.silveira@gmail.com.
- Adriana Sadowski de Souza, Especialista em Tecnologias Aplicadas a Sistemas de Informação com Métodos Ágeis (UniRitter – Centro Universitário Ritter dos Reis), adrianasadowski@gmail.com.